LecChip(レクチンマイクロアレイ)のデータを使用して、糖鎖の構造、細胞種などを判別させるためには、Deep Learningを使用して沢山のデータを機械学習させる方法が有効です。
これを行う為に必要な事前準備は、以下となります。
Python(以下ではAnaconda3を使用)
Tensorflow
Keras
先ずは、ご自身のパソコンにこれらをインストールして環境構築を行ってください。
このような事前準備の面倒臭さを省き、Deep Learningのネットワーク構成をマウスでのクリック動作だけで出来るようにしたのが弊社製品の「SA/DL Easy」になります。
SA/DL Easyを使うと、環境構築の必要もなく、以下のようなScriptも書かずに、Deep Learningの世界がたやすく使えるようになります。
下記を自身のパソコンで実行される場合には、PythonのScriptを保存したフォルダー内にpathが通っていること、保存した入力データのpath、学習結果とテスト結果が保存されているフォルダーのpathなどを間違えないように指定してください。
———————————————————————————————
# LecChipのデータを使って、例えば、糖鎖の構造や細胞種の判断を学習させるためのDeep Learning Python Script例
from __future__ import print_function
import numpy as np
import csv
import pandas
from keras.datasets import mnist
from keras.models import Sequential
from keras.layers.core import Dense, Dropout, Activation
from keras.optimizers import RMSprop
from keras.utils import np_utils
from make_tensorboard import make_tensorboard
np.random.seed(1671) # 再現性を良くするために
# ニューラルネットワークの構成と学習のさせ方
NB_EPOCH = 100 # 何回学習させるか、適当に決めてください
BATCH_SIZE = 2 # データセットを幾つかのサブセットに分ける
VERBOSE = 1
NB_CLASSES = 2 # 最終的な出力数
OPTIMIZER = RMSprop() # オプティマイザー
N_HIDDEN = 45 # 隠れ層のノード数、ここではLecChipのレクチン数に合わせて45としている
VALIDATION_SPLIT = 0.2 # 学習データの内、何割をテストデータとして使うか
DROPOUT = 0.3
LECTINS = 45
def drop(df):
return df[pandas.to_numeric(df.iloc[:, 2], errors=’coerce’).notnull()]
# データは最大値を1とするように規格化する
def normalize_column(d):
dmax = np.max(d)
dmin = np.min(d)
return (np.log10(d + 1.0) – np.log10(dmin + 1.0)) / \
(np.log10(dmax + 1.0) – np.log10(dmin + 1.0))
def normalize(data):
return np.apply_along_axis(normalize_column, 0, data)
# 入力するデータはCSVファイルの形式とする
df1 = drop(pandas.read_csv(r’c:\Users\Masao\Anaconda3\DL_scripts\cell.csv’)).reset_index(drop=True)
X_train = normalize(df1.iloc[:, 2:].astype(np.float64))
family_column = df1.iloc[:, 1]
family_list = sorted(list(set(family_column)))
Y_train = np.array([family_list.index(f) for f in family_column])
df2 = drop(pandas.read_csv(r’c:\Users\Masao\Anaconda3\DL_scripts\cell_test.csv’)).reset_index(drop=True)
X_test = normalize(df2.iloc[:, 2:].astype(np.float64))
familyt_column = df2.iloc[:, 1]
familyt_list = sorted(list(set(familyt_column)))
Y_test = np.array([familyt_list.index(f) for f in familyt_column])
print(X_train.shape[0], ‘train samples’)
print(X_test.shape[0], ‘test samples’)
# convert class vectors to binary class matrices
Y_train = np_utils.to_categorical(Y_train, NB_CLASSES)
Y_test = np_utils.to_categorical(Y_test, NB_CLASSES)
print(X_train)
print(Y_train)
print(X_test)
print(Y_test)
# ニューラルネットワークの具体的な構成例
# 隠れ層は2層
# 入力はLecChipのデータ(45レクチンを使用)
# 最終層はsoftmaxで活性化
model = Sequential()
model.add(Dense(N_HIDDEN, input_shape=(LECTINS,)))
model.add(Activation(‘relu’))
model.add(Dropout(DROPOUT))
model.add(Dense(N_HIDDEN))
model.add(Activation(‘relu’))
model.add(Dropout(DROPOUT))
model.add(Dense(NB_CLASSES))
model.add(Activation(‘softmax’))
model.summary()
# 学習とテスト結果状況をTensorboardに出力して可視化させる
callbacks = [make_tensorboard(set_dir_name=’Glycan_Profile’)]
model.compile(loss=’categorical_crossentropy’,
optimizer=OPTIMIZER,
metrics=[‘accuracy’])
model.fit(X_train, Y_train,
batch_size=BATCH_SIZE, epochs=NB_EPOCH,
callbacks=callbacks,
verbose=VERBOSE, validation_split=VALIDATION_SPLIT)
score = model.evaluate(X_test, Y_test, verbose=VERBOSE)
print(“\nTest score:”, score[0])
print(‘Test accuracy:’, score[1])
————————————————————————————
# Tensorboardを使うためのPython Script
# -*- coding: utf-8 -*-
from __future__ import absolute_import
from __future__ import unicode_literals
from time import gmtime, strftime
from keras.callbacks import TensorBoard
import os
def make_tensorboard(set_dir_name=”):
ymdt = strftime(“%a_%d_%b_%Y_%H_%M_%S”, gmtime())
directory_name = ymdt
log_dir = set_dir_name + ‘_’ + directory_name
os.mkdir(log_dir)
tensorboard = TensorBoard(log_dir=log_dir, write_graph=True, )
return tensorboard
————————————————————————————
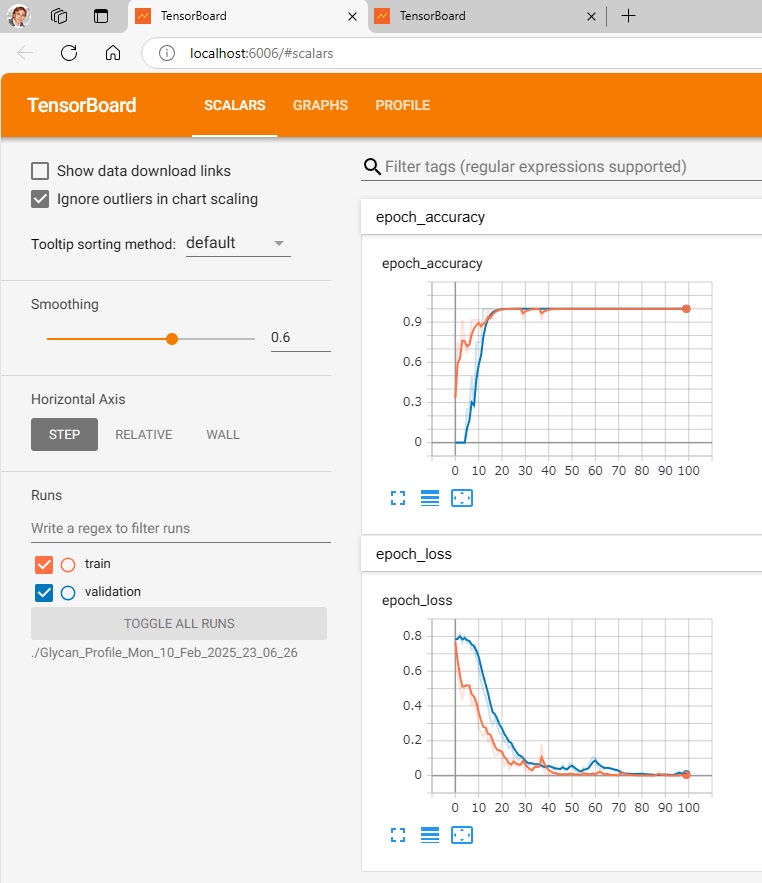
Tensorboardを用いて可視化させるには、make_tensorboard.pyを走らせたうえで、
$ tensorboard –logdir=./Glycan_Profile_Mon_10_Feb_2025_23_06_26(./データが記録されているフォルダー)を走らせ
http://localhost:6006/にアクセスします。
(base) PS C:\Users\masao\Anaconda3\DL_Scripts> python make_tensorboard.py
Using TensorFlow backend.
(base) PS C:\Users\masao\Anaconda3\DL_Scripts> tensorboard –logdir=./Glycan_Profile_Mon_10_Feb_2025_23_06_26
Serving TensorBoard on localhost; to expose to the network, use a proxy or pass –bind_all
TensorBoard 2.0.2 at http://localhost:6006/ (Press CTRL+C to quit)
—————————————————————————————————-
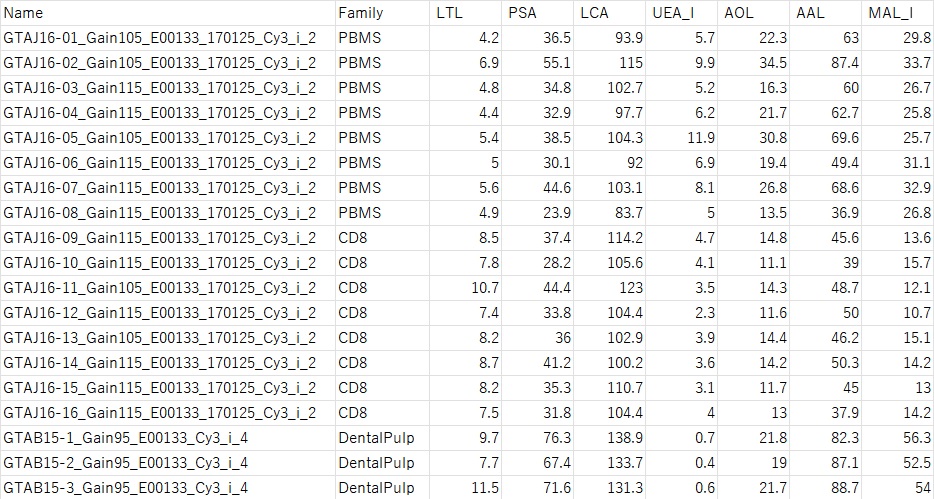
LecChipのデータは、下記のようなCSV形式とします。
左端から、サンプル名、ファミリー名(教師データとなります)、各種レクチンの数値が並んでいます。
—————————————————————————————————-
学習が終わったら、モデルの保存をしておくべきでしょう。
モデルが保存してあれば復元もできますし、未知データを与えて予測させることが出来ます。
この辺りのScriptは別途書くことにします。